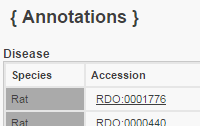
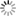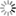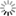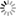Imported Disease Annotations - CTDObject Symbol | Species | Term | Qualifier | Evidence | With | Reference | Notes | Source | Original Reference(s) | AP3S2 | Human | type 2 diabetes mellitus | | EXP | | 11554173 | CTD Direct Evidence: marker/mechanism | CTD | PMID:21874001 | | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Gene-Chemical Interaction Annotations Click to see Annotation Detail View
Gene Ontology Annotations Click to see Annotation Detail View
Biological Process
Cellular Component