Imported Disease Annotations - CTDObject Symbol | Species | Term | Qualifier | Evidence | With | Reference | Notes | Source | Original Reference(s) | WDR20 | Human | papillary thyroid carcinoma | | EXP | | 11554173 | CTD Direct Evidence: marker/mechanism | CTD | PMID:28030816 | | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|
|
Gene-Chemical Interaction Annotations Click to see Annotation Detail View
Gene Ontology Annotations Click to see Annotation Detail View
Cellular Component
Molecular Function
Phenotype Annotations Click to see Annotation Detail View
Imported Human Phenotype Annotations - ClinVar
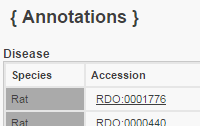
Disease Annotations
Click to see Annotation Detail View
|
|
Gene-Chemical Interaction Annotations
Click to see Annotation Detail View
|
|
|
Gene Ontology Annotations
Click to see Annotation Detail View
Phenotype Annotations
Click to see Annotation Detail View
References
References - curated
| # | Reference Title | Reference Citation |
| 1. | Data Import for Chemical-Gene Interactions | RGD automated import pipeline for gene-chemical interactions |
Additional References at PubMed
| PMID:8125298 | PMID:8889548 | PMID:12477932 | PMID:14702039 | PMID:15342556 | PMID:15489334 | PMID:17081983 | PMID:18029348 | PMID:18925368 | PMID:19615732 | PMID:19913121 | PMID:20147737 |
| PMID:20628086 | PMID:21183687 | PMID:22990118 | PMID:23444366 | PMID:24356955 | PMID:24705354 | PMID:24981860 | PMID:25216524 | PMID:25814554 | PMID:25852190 | PMID:26186194 | PMID:26462181 |
| PMID:26496610 | PMID:26638075 | PMID:26673895 | PMID:26760575 | PMID:26790128 | PMID:26811477 | PMID:27173435 | PMID:27373336 | PMID:27463890 | PMID:27880917 | PMID:28514442 | PMID:28883622 |
| PMID:28986522 | PMID:29568061 | PMID:29655804 | PMID:30021884 | PMID:30194290 | PMID:30466959 | PMID:30639242 | PMID:30945288 | PMID:31266804 | PMID:31527615 | PMID:31753913 | PMID:32296183 |
| PMID:32694731 | PMID:32814053 | PMID:33397691 | PMID:33844468 | PMID:33961781 | PMID:34079125 | PMID:34369648 | PMID:35140242 | PMID:35271311 | PMID:35337019 | PMID:35844135 | PMID:36215168 |
| PMID:36976175 | PMID:37289831 | PMID:37433992 | PMID:38117590 | PMID:39432777 |
Genomics
Comparative Map Data
| WDR20 (Homo sapiens - human) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Wdr20 (Mus musculus - house mouse) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Wdr20 (Rattus norvegicus - Norway rat) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Wdr20 (Chinchilla lanigera - long-tailed chinchilla) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WDR20 (Pan paniscus - bonobo/pygmy chimpanzee) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WDR20 (Canis lupus familiaris - dog) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Wdr20 (Ictidomys tridecemlineatus - thirteen-lined ground squirrel) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WDR20 (Sus scrofa - pig) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| WDR20 (Chlorocebus sabaeus - green monkey) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Wdr20 (Heterocephalus glaber - naked mole-rat) |
|
||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Variants
.
Variants in WDR20
31 total Variants 
|